Mittelwertvergleich
Diese Einheit behandelt den Mittelwertvergleich. Dieser bietet den Rahmen für die Analyse des Zusammenhangs zwischen einer kategorialen (bzw. nicht-metrischen) unabhängigen und einer kontinuierlichen (bzw. metrischen) abhängigen Variable. Wir arbeiten wiederum mit den Befragungsdaten aus dem Kurs (kursdata_anon).
# Sitzung 10 Mittelwertvergleiche
# Statistik 1: R Tutorat
# Übungsskript zum Mittelwertvergleich
# Datum: 20.12.2023
# AutorIn: XXX
Vorbereitung: Einlesen der Daten und Aktivieren der nötigen Packages
# Working directory setzen (z.B. "c:\daten")
setwd("mein_laufwerk/mein_datenverzeichnis")
# Daten einlesen
library(haven)
kursdata_anon <- read_dta("kursdata_anon.dta")#install.packages("tidyverse")
library(tidyverse)
#install.packages("ggplot2")
library(ggplot2)
1 Vorbereitung und Dateninspektion
Wir wollen den Zusammenhang zwischen den Variablen sidejob und lezufr analysieren. Wir vermuten, dass für Studierende die Ausübung eines Nebenjobs einen Einfluss auf die Lebenszufriedenheit hat.
Zuerst verschaffen wir uns mit den Befehlen attributes, summary und table einen Überblick über die beiden relevanten Variablen:
attributes(kursdata_anon$sidejob)## $label
## [1] "sind sie neben dem studium erwerbstätig"
##
## $format.stata
## [1] "%8.0g"
##
## $class
## [1] "haven_labelled" "vctrs_vctr" "double"
##
## $labels
## nein ja
## 1 2table(kursdata_anon$sidejob)##
## 1 2
## 21 53sidejob ist eine kategoriale Variable mit zwei Ausprägungen. Die Mehrheit im Kurs (53 Personen) übt neben dem Studium einen Nebenjob aus. Die Variable ist im Datensatz als numerische Variable mit den Kategorien 1 und 2 angelegt, zur besseren analytischen Einbindung faktorisieren wir sie mit as_factor:
kursdata_anon$sidejob <- as_factor(kursdata_anon$sidejob)
table (kursdata_anon$sidejob)##
## nein ja
## 21 53Gelegentlich kommt es durch Faktorisierungen an dieser Stelle zur Einbindung leerer Phantomkategorien in die Variable (siehe z.B. letzte Einheit). Dieses ist hier aber, ausweislich der Häufigkeitsauszählung oben, nicht der Fall. Z ur Lebenszufriedenheit:
attributes(kursdata_anon$lezufr)## $label
## [1] "Lebenszufriedenheit derzeit"
##
## $format.stata
## [1] "%8.0g"summary(kursdata_anon$lezufr)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## -99.00 60.00 75.00 65.53 80.00 100.00Unsere AV lezufr ist eine metrische Variable, welche die Lebenszufriedenheit zum Zeitpunkt der Befragung auf einer Skala von 0 bis 100 misst. Die Missingkategorie -99 wurde nicht korrekt als NA rekodiert, weshalb wir dies nun manuell nachholen:
kursdata_anon$lezufr[kursdata_anon$lezufr == -99] <- NA
summary(kursdata_anon$lezufr)## Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
## 0.00 61.50 75.00 67.72 80.00 100.00 1sd(kursdata_anon$lezufr, na.rm = TRUE)## [1] 21.046
2 Einfacher Mittelwertvergleich: Gruppenspezifische Analysen
Um den Einfluss bzw. die Einflussgrösse eines Nebenjobs auf die Lebenszufriedenheit zu messen, vergleichen wir die durchschnittliche Lebenszufriedenheit der Nebenjobber mit der durchschnittlichen Lebenszufriedenheit der Nicht-Nebenjobber. Den Unterschied der beiden gruppenspezifischen Zufriedenheitswerte interpretieren wir dann als Einfluss eines Nebenjobs auf die Lebenszufriedenheit - versehen mit dem Disclaimer, dass wir die Kausalrichtung nicht statistisch, sondern annahmegestützt einbinden.
In R setzen wir dies um, indem wir zunächst zwei getrennte Datensätze erstellen, die je eine der beiden Ausprägungensgruppen der UV enthalten: Erwerbstätige und nicht-Erwerbstätige Studierende.
library(dplyr)
work <- filter(kursdata_anon, sidejob == "ja")
nowork <- filter(kursdata_anon, sidejob == "nein")Die Werteliste der Lebenszufriedenheit ist nun aufgesplittet nach Jobstatus in den beiden Teildatensätzen ausgewiesen. Mit summary()- oder mean- können wir nun also die lezufr-Mittelwerte der beiden Teilgruppen miteinander vergleichen.
mean(work$lezufr, na.rm = TRUE)## [1] 69.01923mean(nowork$lezufr, na.rm = TRUE)## [1] 63.33333Alternative Umsetzung ohne Teildatensatzbildung, dafür mit datensatzintegrierter Gruppenbildung:
kursdata_anon_gr<-group_by(kursdata_anon, sidejob)
summarise_at(kursdata_anon_gr, vars(lezufr), list(mean=mean, sd=sd), na.rm=TRUE)## # A tibble: 3 × 3
## sidejob mean sd
## <fct> <dbl> <dbl>
## 1 nein 63.3 25.7
## 2 ja 69.0 19.2
## 3 <NA> 80 2.83Tatsächlich finden wir einen Unterschied der Lebenszufriedenheit zwischen den beiden Gruppen: Die mittlere Lebenszufriedenheit der arbeitenden Studierenden liegt bei 69.0, die der nicht arbeitenden Studierenden bei 63.3. Die durchschnittliche Lebenszufriedenheit arbeitender Studierender liegt also 5.7 Skalenpunkte (Mittelwertdifferenz) oberhalb der von nicht arbeitenden Studierenden.
Die Mittelwertdifferenz überspannt einen mittelgrossen Abschnitt der
100er-Lebenszufriedenheitsskala; die meisten von uns würden, gestützt
auf den eigenen Zugang zur Skala, einem Unterschied von 5.7
Skalenpunkten wahrscheinlich eine gewisse substanzielle Bedeutung
zumessen. Er beträgt zudem immerhin 5.7/21=0,27 Einheiten
der Standardabweichung (und somit mehr als ein Viertel einer typischen
Abweichung in der Lebenszufriedenheit), was ebenfalls für eine
Einordnung als inhaltlich bedeutsamer Unterschied bzw. kleiner bis
mittelerer Effekt spricht.
Unklar ist auf Basis unserer Analyse bisher gleichwohl,
- ob die Mittelwertdifferenz auch statistisch signifikant von 0 verschieden ist
- mit welcher Abweichung zur Differenz in der Population (aller Soziologiestudierender) wir rechnen müssen
Um solche inferenzstatistische Informationen zu erhalten, ermitteln wir die Mittelwertdifferenz im Rahmen eines t-tests bzw. mit dem R-Kommando t.test
3 Mittelwertvergleich mit t.test
Wir können die Mittelwerte der Teilgruppen auch ohne Erstellung von zwei Teildatensätzen direkt mit dem Befehl t.test() ermitteln. Dieses Kommando nimmt ausserdem inferenzstatistische Berechnungen (Konfidenzintervall, p-Wert) vor. R kennt zwei grundsätzliche Spezifikationsmöglichkeiten des t-tests:
# Differenzierung der Gruppen nach Kategorien der UV innerhalb des Kommandos
t.test(formula = lezufr ~ sidejob, var.equal = TRUE, data = kursdata_anon)##
## Two Sample t-test
##
## data: lezufr by sidejob
## t = -1.0363, df = 71, p-value = 0.3036
## alternative hypothesis: true difference in means between group nein and group ja is not equal to 0
## 95 percent confidence interval:
## -16.62656 5.25476
## sample estimates:
## mean in group nein mean in group ja
## 63.33333 69.01923# Differenzierung der Gruppen nach Kategorien der UV ausserhalb des Kommandos
t.test(nowork$lezufr, work$lezufr, var.equal = TRUE)##
## Two Sample t-test
##
## data: nowork$lezufr and work$lezufr
## t = -1.0363, df = 71, p-value = 0.3036
## alternative hypothesis: true difference in means is not equal to 0
## 95 percent confidence interval:
## -16.62656 5.25476
## sample estimates:
## mean of x mean of y
## 63.33333 69.01923Mit formula = AV ~ UV spezifizieren wir in der
ersten Variante, welcher Zusammenhang geprüft werden soll - und machen
ausserdem automatisch klar, dass wir es mit ungepaarten
Beobachtungen zu tun haben (die Struktur des Befehls unterscheidet
sich für Vergleiche gepaarter Beobachtungen, siehe
? t.test). Alternativ kann (zweite Variante) derselbe
t-test auch durch direkte Spezifikation der beiden Werterlisten, deren
Mittelwerte verglichen werden sollen, umgesetzt werden. Mit
var.equal = TRUE legen wir in beiden Varianten fest,
dass die Standardversion des t-tests durchgeführt werden soll (bei der
von Varianzhomogenität zwischen den beiden Teilgruppen ausgegangen
wird).
Im Output unter sample estimates präsentiert uns R die zuvor auch manuell berechneten Mittelwerte: 63.3 vs. 69.0. Die sich daraus ergebende Mittelwertdifferenz ist nicht Teil des Outputs, daher machen wir Sie manuell sichtbar:
58.5-70.5## [1] -12Achtung: R betrachtet die Mittelwertdifferenz nicht als Betrag, sondern als Tendenz bzw. als Differenz von der zweiten zur ersten Gruppe. Die Mittelwertdifferenz hat daher ein Vorzeichen: In der ersten Gruppe, den Nicht-Nebenjobbern, ist der Mittelwert 5.7 Skalenpunkte kleiner als in der zweiten Gruppe, den Nebenjobbern.
Der Standardfehler der Mittelwertdifferenz ist nicht mit im Standardoutput zu t.test dargestellt, ist aber ebenfalls ein wichtiger inferenzstatistischer Parameter des Mittelwertvergleichs. Er kann gezielt durch eine einfache Erweiterung des Kommandos aufgedeckt werden:
t.test(formula = lezufr ~ sidejob, var.equal = TRUE, data = kursdata_anon)$stderr## [1] 5.486945…wir müssen also erwarten, dass die in der Stichprobe gefundene Mittelwertdifferenz etwa 5,5 Skalenpunkte von der wahren Mittelwertdifferenz in der Population abweicht.
Die ausgewiesenen Grenzen des
95%-Konfidenzintervalls ergeben sich, wie ihr leicht
nachrechnen könnt, näherungsweise aus empirischer Mittelwertdifferenz
und Standardfehler [-5.7 ± 5,5*1,96]. Es zeigt die Grenzen
des Intervalls an, in dem sich die «wahre» Mittelwertdifferenz der
Populationen mit 95%-Sicherheit befindet. Mit 95% Sicherheit liegt
die durchschnittliche Lebenszufriedenheit nicht-arbeitender Studierender
in der Population also zwischen 16,6 Skalenpunkten unterhalb und 5,3
Punkten oberhalb der durchschnittlichen Lebenszufriedenheit arbeitender
Studierender.
Zusätzlich berechnet der t.test()-Befehl den p-Wert für den empirischen Mittelwertunterschied. Der hier ermittelte p-Wert von 0.30 zeigt an, dass die Wahrscheinlichkeit eines Zufallsergebnisses (genauer: die Wahrscheinlichkeit, dass eine Grundgesamtheit ohne Mittelwertunterschied eine Stichprobe mit dem gefunden oder grösseren Mittelwertunterschied erzeugt) relativ gross ist und konkret bei etwa 30% liegt. Ein p-Wert kleiner als 0.05 (manchmal auch 0.01 oder 0.1) wird in der Regel als statistisch signifikant bewertet. Auf dem 5% Signifikanzniveau kann eine entsprechende Nullhypothese also nicht abgelehnt werden.
Der t.test.()-Befehl berechnet standardmässig den
p-Wert eines zweiseitigen Hypothesentests. Durch Modifikation des
Befehls kann aber optional auch die Teststatistik eines einseitigen
Hypothesentest angefordert werden (siehe ?t.test). Diese
erhalten wir gleichwohl auch durch einfache Division des zweiseitigen
p-Wertes durch 2 (vorausgesetzt, die Tendenz zwischen
Forschungshypothese und empirischem Unterschied stimmt überein).
4 Visualisierung von Mittelwertvergleichen
Mit dem ggplot()-Befehl (spezifisch dem geom_boxplot()-Teilbefehl) lassen sich Mittelwerte sowie weitere Verteilungsmerkmale (Range, Quartilsgrenzen) auch in vergleichender Perspektive visualisieren. Zuerst betrachten wir den Teildatensatz “work”:
library(ggplot2)
box_work <- ggplot(work, aes(y = lezufr, x = sidejob)) +
geom_boxplot(colour = "darkblue", fill = "lightpink") +
labs(x = "", y = "Lebenszufriedenheit",
title = "Verteilung Lebenszufriedenheit bei Studierenden mit Nebenjob",
subtitle = "Wie zufrieden bist du mit deinem Leben auf einer Skala
von 1 (gar nicht) bis 100 (sehr)?",
caption = "Quelle: Kursbefragung Statistik I, Teildatensatz") +
scale_x_discrete(label=c("")) +
stat_summary(fun=mean, geom="point", shape=23, size=3, color="magenta3", fill="white") +
ylim(0, 100) +
theme_bw()
box_work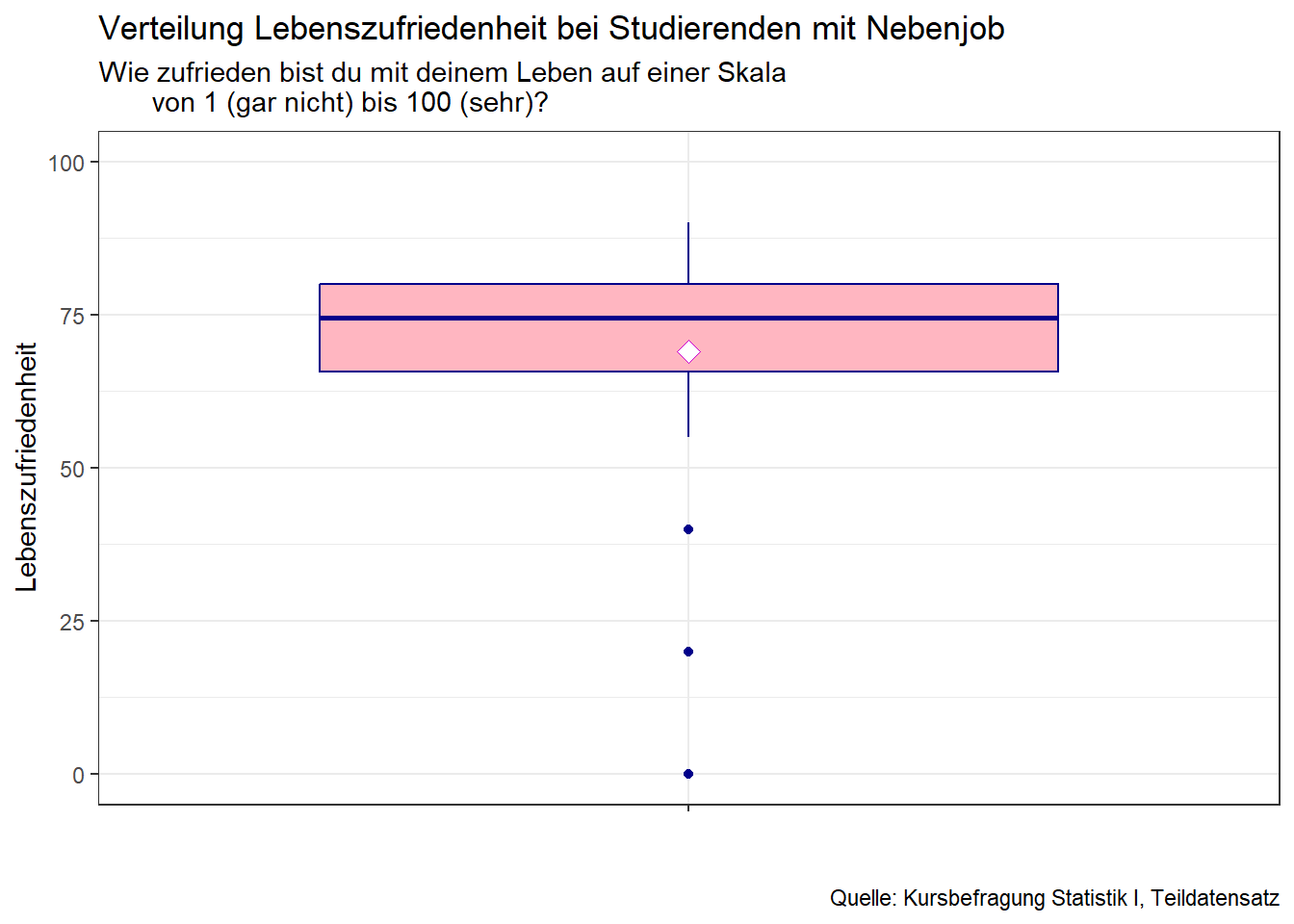
Hier sehen wir die univariate Verteilung des Teildatensatzes “work”
(Vgl. Kapitel IV: Univariate Statistik.) Die Linie innerhalb des
Boxplots stellt den Median dar (Achtung: in der
klassischen Variante des Boxplots nicht das arithmetisches Mittel), die
Aussengrenzen bilden das 1. bzw. 3. Quartil ab. Darüber hinaus bilden
die von dem Zentrum des Boxplots ausgehenden Striche an ihren Enden
jeweils die Grenzen der Kernverteilung ab. Zum Schluss lassen sich drei
Ausreisserwerte erkennen. Mit stat_summary fügen wir
ausserdem die weisse Raute, welche das arithmetische Mittel zeigt,
ein.
Nun können wir dieselbe Visualisierung für den Teildatensatz “nowork”
erstellen:
box_nowork <- ggplot(nowork, aes(y = lezufr, x = sidejob)) +
geom_boxplot(colour = "darkblue", fill = "lightpink") +
labs(x = "", y = "Lebenszufriedenheit",
title = "Verteilung Lebenszufriedenheit bei Studierenden ohne Nebenjob",
subtitle = "Wie zufrieden bist du mit deinem Leben auf einer Skala
von 1 (gar nicht) bis 100 (sehr)?",
caption = "Quelle: Kursbefragung Statistik I, Teildatensatz") +
scale_x_discrete(label=c("")) +
stat_summary(fun=mean, geom="point", shape=23, size=3, color="magenta3", fill="white") +
ylim(0, 100) +
theme_bw()
box_nowork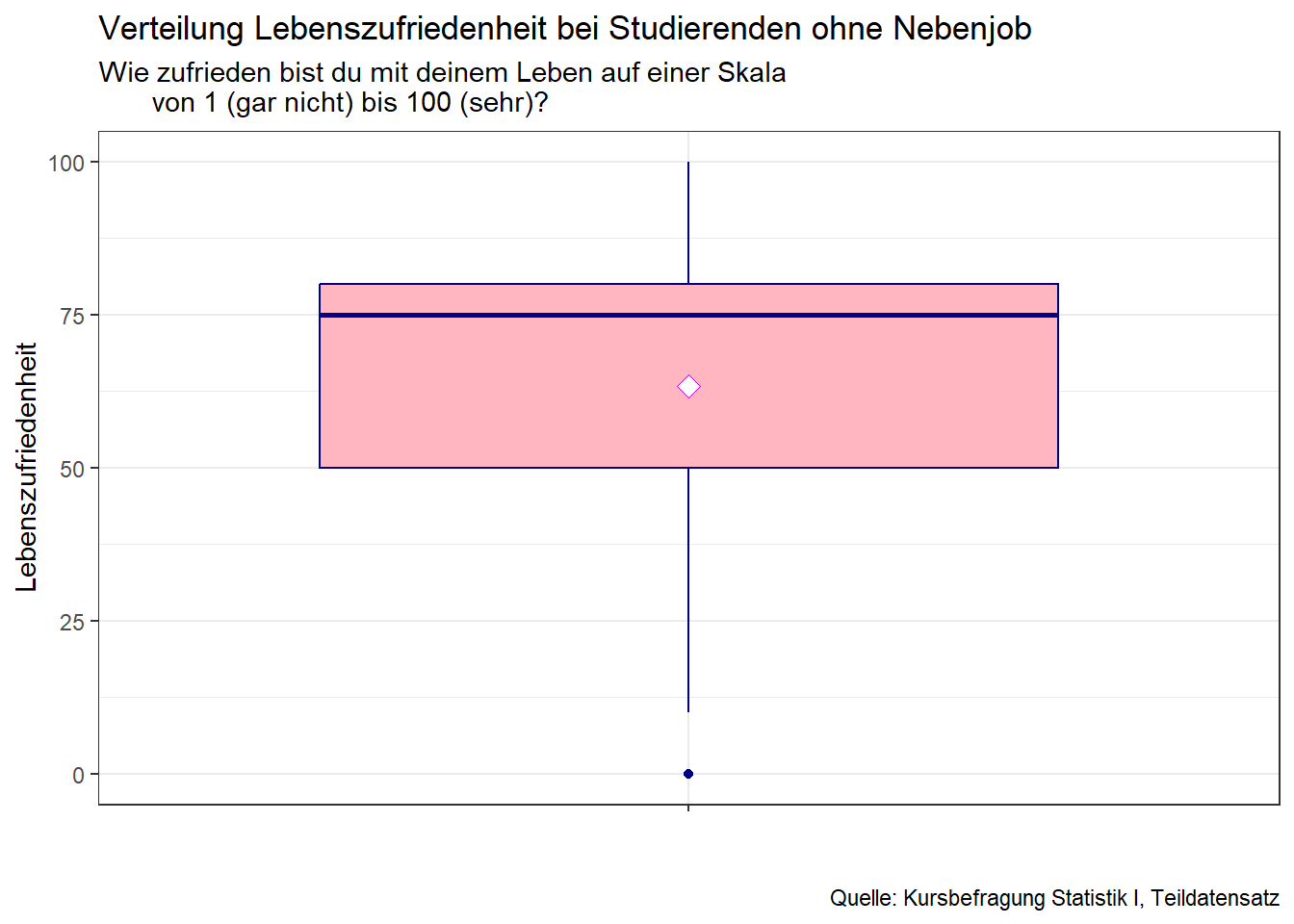
Obwohl die beiden Plots den gleichen Zusammenhang visualisieren (UV:
Nebenjob, AV: Lebenszufriedenheit), können wir die beiden Outputs schwer
miteinander vergleichen, da sie sich in anderen Export-Dateien befinden.
Im ggplot-Befehl können wir beide Teildatensätze
allerdings integriert bzw. vergleichend nebeneinander
darstellen, indem wir sowohl x als auch y im Befehl spezifizieren und
die beiden Kategorien von x vergleichen.
Hinweis: Der ggplot-Befehl hat allerdings
einen entscheidenden Nachteil: Die Kategorie “keine Angabe” (also unsere
NA-Werte) werden nicht automatisch aus der Grafik gelöscht,
sondern wird mit angezeigt. Deshalb erstellen wir zuerst einen
Teildatensatz mit den für die Visualisierung relevanten Variablen und
entfernen anschliessend alle NA’s mit dem
na.omit-Befehl. Alternativ könnten wir im Rahmen eines
einzelnen Kommandos mit dem Filter-Befehl arbeiten -
siehe vorhergehende Lerneinheit.
library(tidyverse)
subset <- select(kursdata_anon, sidejob, lezufr)
clean <- na.omit(subset)box <- ggplot(clean,
aes(y = lezufr, x = sidejob)) +
geom_boxplot(colour = "darkblue", fill = "lightpink") +
labs(y = "Lebenszufriedenheit",
x = "Nebenjob",
title = "Verteilung Lebenszufriedenheit nach Nebenjob",
subtitle = "Wie zufrieden bist du mit deinem Leben auf einer Skala
von 1 (gar nicht) bis 100 (sehr)?",
caption = "Quelle: Kursbefragung Statistik I") +
ylim(0, 100) +
stat_summary(fun=mean, geom="point", shape=23, size=3, color="magenta3", fill="white") +
theme_bw()
box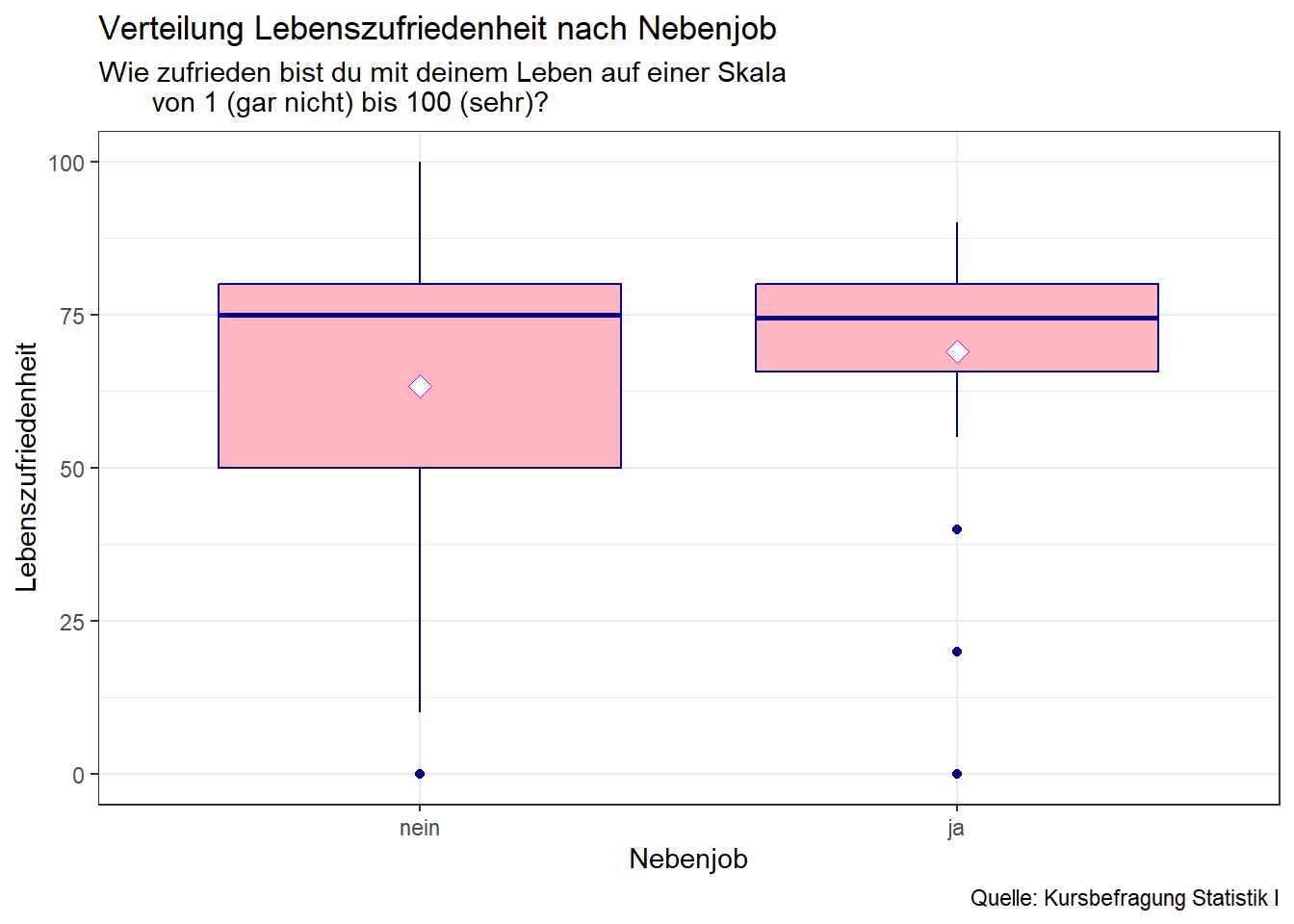
Alternativ lässt sich der Zusammenhang zwischen Nebenjob und der Lebenszufriedenheit in einem Histogramm oder Violinplot darstellen, welche wir bereits in der univariaten Statistik kennengelernt haben.
hist <- ggplot(clean,
aes(x = lezufr, fill = sidejob)) +
geom_histogram(alpha=0.3, position="identity", bins = 10,
breaks = c(0,10,20,30,40,50,60,70,80,90,100)) +
labs(x = "Lebenszufriedenheit",
y = "Anzahl",
title = "Verteilung Lebenszufriedenheit nach Nebenjob",
subtitle = "Wie zufrieden bist du mit deinem Leben auf einer Skala
von 1 (gar nicht) bis 100 (sehr)?",
fill = "Nebenjob",
caption = "Quelle: Kursbefragung Statistik I") +
scale_x_continuous(breaks = seq(0,100,10)) +
scale_y_continuous(breaks = seq(0,30,2)) +
theme_bw()
hist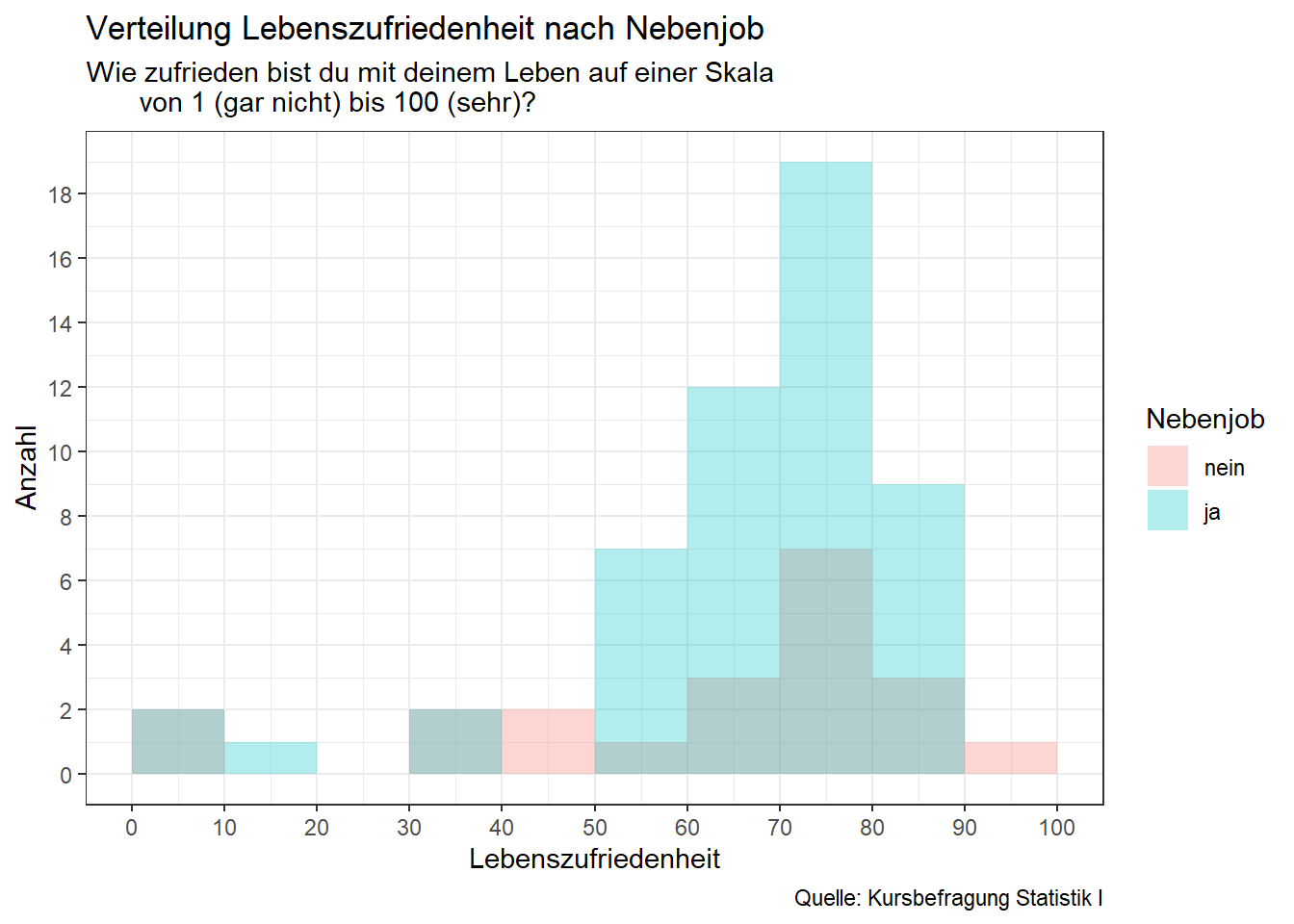
Mithilfe des geom_histogram() Teilbefehls lassen sich zwei übereinandergelegte Histogramme plotten, welche uns die UV-ausprägungsspezifischen Verteilungen der AV aufzeigen. Beachte, dass wir über die Spezifizerung eines alpha die Transparenz des Histogramms bestimmen. Hier können Werte zwischen 0 (komplett transparent) bis 1 (nicht transparent) gewählt werden.
violin <- ggplot(clean,
aes(y = lezufr, x = sidejob)) +
geom_violin(colour = "darkblue", fill = "lightpink") +
labs(y = "Lebenszufriedenheit",
x = "Nebenjob",
title = "Verteilung: Lebenszufriedenheit nach Nebenjob",
caption = "Quelle: Kursbefragung Statistik I (n = 73)") +
ylim(0, 100) +
stat_summary(fun=mean, geom="point", shape=23, size=2, color="magenta3", fill="white") +
theme_bw()
violin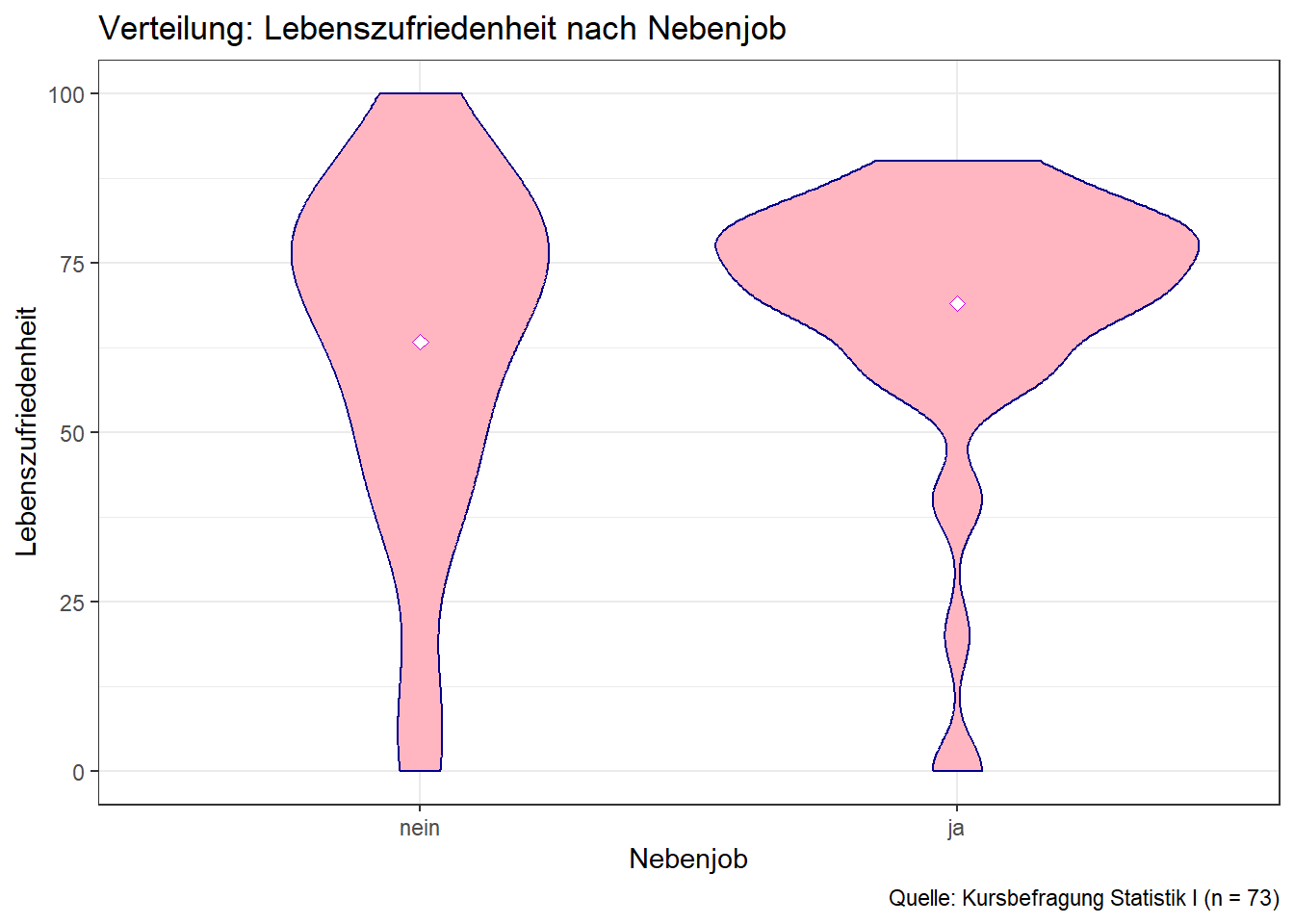
box <- ggplot(clean,
aes(y = lezufr, x = sidejob, fill=sidejob)) +
geom_boxplot (outlier.shape = NA)+
labs(y = "Lebenszufriedenheit",
x = "Nebenjob",
title = "Verteilung: Lebenszufriedenheit nach Nebenjob",
caption = "Quelle: Kursbefragung Statistik I") +
ylim(0, 100) +
stat_summary(fun=mean, geom="point", shape=23, size=3, color="magenta3", fill="white") +
theme(legend.position="none") +
geom_jitter(alpha=0.5, position=position_jitter(0.08))
theme_bw()
box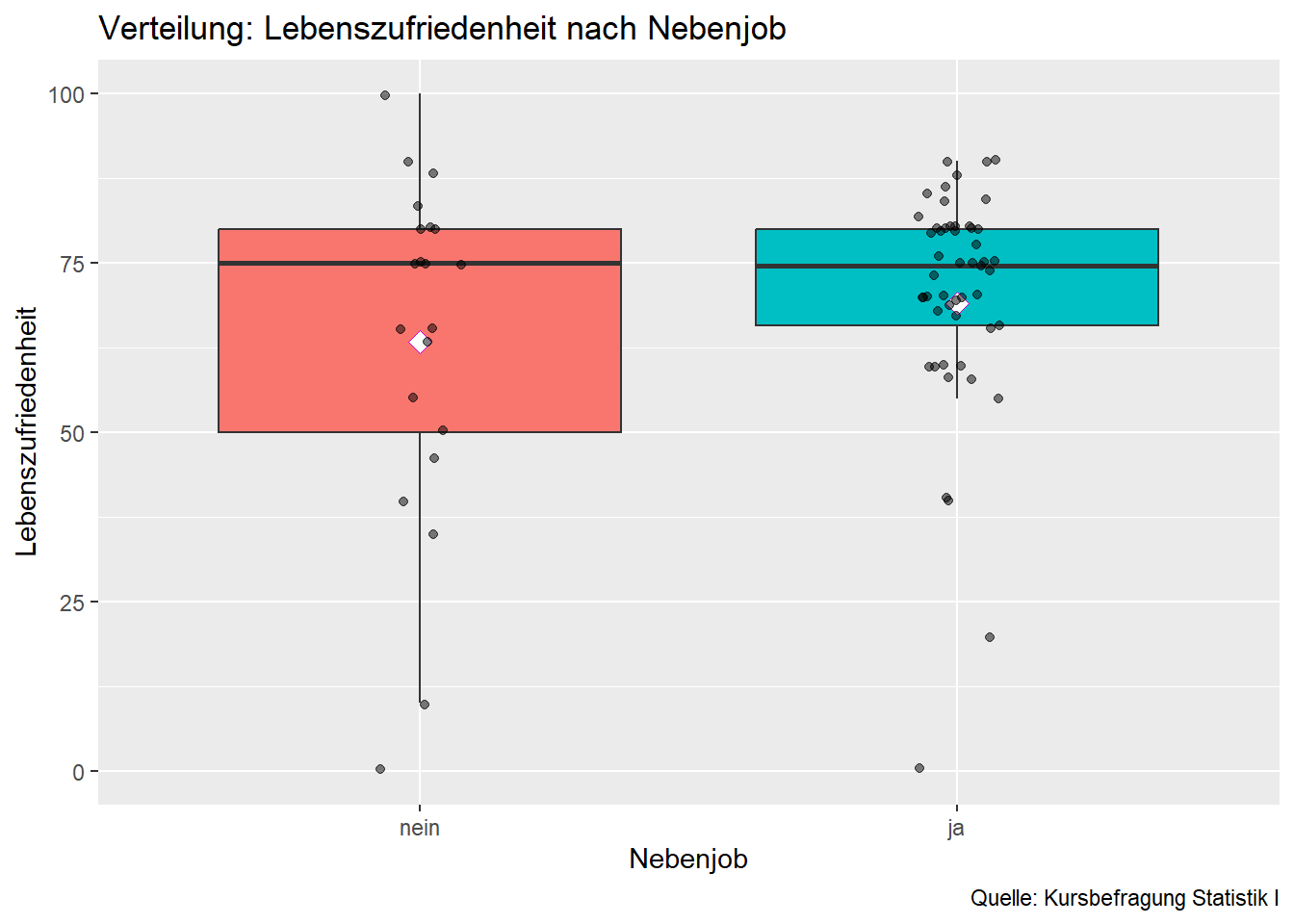
…hier wurde zuletzt nun noch die Funktionalität des Boxplots mit der eines sog. Jitterplots kombiniert, indem der differenzierte Boxplot mit den kategorienspezifischen Punktwolken angereichert wird. Achtung: Die zusätzlichen Parameter im Befehl oben müssen auf jede neue empirische Anwendung hin neu ausgerichtet werden, sonst kommen zumeist unsinnige Darstellungen dabei raus.
Folgende Texte empfehlen wir euch zur Lektüre und Vertiefung:
BCP Kapitel 5: Beckerman, A.P., Childs, D.Z., Petchey, O.L. (2017): Getting Started with R. Oxford: University Press.
SVS Kapitel 9.4: Marco R. Steenbergen, Kushtrim Veseli, Benjamin Schlegel (2015): Working with Descriptive and Inferential Statistics in R. Script.